Thème de recherche n°1 : Mécanismes de régulation de l'expression génique par des ARNs
La régulation de l'expression des gènes joue un rôle clé dans l’adaptation des bactéries à leur environnement. Cette régulation a d'abord été mise en évidence au niveau de la transcription, la première étape de l'expression génique. Ce n’est que plus tard que la régulation à des étapes postérieures à la transcription, également appelée contrôle post-transcriptionnel, a été découverte. Le rôle crucial que joue l'ARN dans le contrôle post-transcriptionnel chez les bactéries, mais aussi dans les autres domaines du vivant, est connu depuis plusieurs décennies. Depuis une quinzaine d'années cependant, le nombre d'ARN régulateurs identifiés a explosé et notre compréhension de la diversité des mécanismes utilisés par ces molécules pour contrôler l'expression génique a fortement progressé.
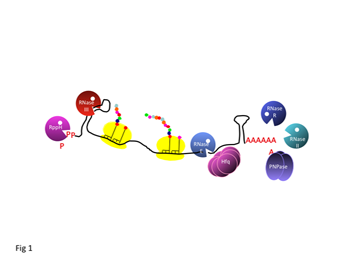
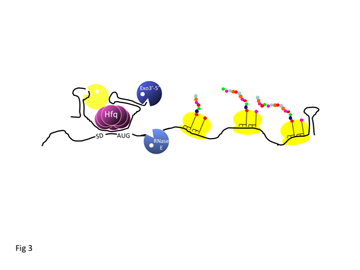
Thème de recherche n°2 : Maturation et stabilité de l'ARN
Chez E. coli, la stabilité des ARN dépend de coupures endoribonucléotidiques qui déclenchent la dégradation des ARN et aussi de l’addition en 3' de séquences poly(A) qui facilitent l’élimination des fragments d’ARN par les exoribonucléases. D’autres facteurs comme les hélicases à ARN, les ribosomes, la protéine Hfq et des ARN régulateurs participent à ces processus. Bien que la majorité des ribonucléases, si ce n'est toutes, qui interviennent dans la maturation ou la dégradation des ARN aient déjà été identifiées, leur fonction spécifique n’est pas connue. Nous avons découvert de manière surprenante que la polyadénylation des ARN bactériens participait à leur dégradation. Longtemps considérée comme une spécificité des ARNm eucaryotes, la polyadénylation est maintenant reconnue comme un processus universel affectant toutes les catégories d’ARN. Chez les eucaryotes et les procaryotes, cette voie de dégradation joue un rôle dans le contrôle de la qualité des ARN en permettant la dégradation spécifique des ARN non fonctionnels. Elle permet également la dégradation exoribonucléolytique des petits fragments d’ARN produits par les endoribonucléases lors de la maturation et l’inactivation des transcrits primaires. De plus, la poly(A)polymérase modifie la stabilité fonctionnelle des ARNm, démontrant ainsi que la polyadénylation peut intervenir dans l’expression génique chez E. coli.
La protéine Hfq est impliquée dans différents mécanismes incluant la dégradation et la traduction. Cette protéine a une fonction essentielle après des stress et participe à l’expression de facteurs de virulence chez de nombreuses bactéries pathogènes. Elle peut agir directement ou indirectement en facilitant l’interaction entre les ARNm et les ARNs. Curieusement, Hfq modifie également l’abondance de certains ARNm et ce indépendamment de leur dégradation. De plus, elle est impliquée dans la dégradation poly(A)dépendante des ARN. Nous mettons en œuvre des approches in vivo et in vitro pour analyser le mode d’action de Hfq ainsi que sa fonction dans le métabolisme cellulaire. Notre objectif principal est ainsi de comprendre comment ces facteurs participent à la régulation de l’expression génique par l’ARN.
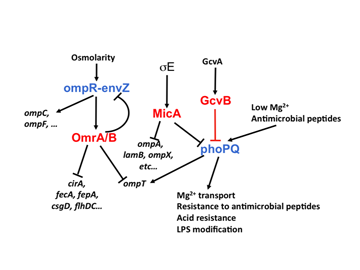
Thème de recherche n°3 : Les ARNs dans les réseaux de régulation de la cellule
De nombreux travaux ont pointé les fortes connexions liant les ARNs à d’autres régulateurs. En effet, si la transcription des ARNs est régulée par des facteurs de transcription ou des facteurs sigma alternatifs, inversement, de nombreux ARNs régulent directement la synthèse de certains facteurs de transcription. La biologie sous-jacente à cette intrication de boucles de régulation est à l'heure actuelle très mal comprise. Nous étudions divers systèmes géniques qui ont fait ressortir cette incroyable et surprenante complexité. Ainsi, deux ARNs indépendants qui régulent par des mécanismes similaires l'expression du système à deux composants PhoP/PhoQ ont des effets totalement différents sur l’expression du régulon PhoP. Si l’on considère la compétition que se livrent les ARNs pour accéder à Hfq et celle à laquelle se livrent plusieurs ARNm pour être ciblés par un même ARNs, nos résultats soulignent la nécessité de faire intervenir la biologie des systèmes dans l'étude de la régulation de l'expression génique par les ARNs.